SLIC (scikit-image に含まれる)のインストールと動作確認(スーパーピクセル)(Python を使用)(Windows 上)
【関連する外部ページ】
スーパーピクセルに関する参考記事: http://scikit-image.org/docs/dev/auto_examples/segmentation/plot_segmentations.html
元画像
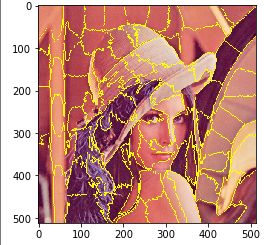
SLIC
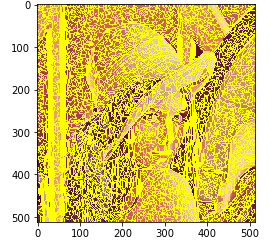
felzenszwalb
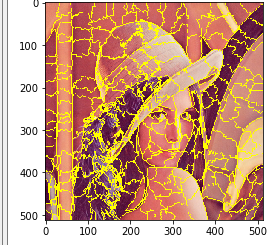
quickshift
watershed
前準備
Python 3.12 のインストール(Windows 上) [クリックして展開]
以下のいずれかの方法で Python 3.12 をインストールする。Python がインストール済みの場合、この手順は不要である。
方法1:winget によるインストール
管理者権限のコマンドプロンプトで以下を実行する。管理者権限のコマンドプロンプトを起動するには、Windows キーまたはスタートメニューから「cmd」と入力し、表示された「コマンドプロンプト」を右クリックして「管理者として実行」を選択する。
winget install --scope machine --id Python.Python.3.12 -e --silent --disable-interactivity --force --accept-source-agreements --accept-package-agreements --override "/quiet InstallAllUsers=1 PrependPath=1 Include_pip=1 Include_test=0 Include_launcher=1 InstallLauncherAllUsers=1"--scope machine を指定することで、システム全体(全ユーザー向け)にインストールされる。このオプションの実行には管理者権限が必要である。インストール完了後、コマンドプロンプトを再起動すると PATH が自動的に設定される。
方法2:インストーラーによるインストール
- Python 公式サイト(https://www.python.org/downloads/)にアクセスし、「Download Python 3.x.x」ボタンから Windows 用インストーラーをダウンロードする。
- ダウンロードしたインストーラーを実行する。
- 初期画面の下部に表示される「Add python.exe to PATH」に必ずチェックを入れてから「Customize installation」を選択する。このチェックを入れ忘れると、コマンドプロンプトから
pythonコマンドを実行できない。 - 「Install Python 3.xx for all users」にチェックを入れ、「Install」をクリックする。
インストールの確認
コマンドプロンプトで以下を実行する。
python --versionバージョン番号(例:Python 3.12.x)が表示されればインストール成功である。「'python' は、内部コマンドまたは外部コマンドとして認識されていません。」と表示される場合は、インストールが正常に完了していない。
SLIC (scikit-image) のインストール
- 以下のコマンドを管理者権限のコマンドプロンプトで実行する
(手順:Windowsキーまたはスタートメニュー →
cmdと入力 → 右クリック → 「管理者として実行」)。
次のコマンドを実行する.
python -m pip install -U scikit-image matplotlib
スーパーピクセルの実行(scikit-image の SLIC,Python を使用)
SLIC
SLIC を行う Python プログラムを実行してみる.
- Python プログラムを実行する..Matplotlib を使うので,Jupyter QtConsole や Jupyter ノートブック (Jupyter Notebook) の利用が便利である.
- Python プログラムを実行する
%matplotlib inline import matplotlib.pyplot as plt import warnings warnings.filterwarnings('ignore') # Suppress Matplotlib warnings import skimage.data import skimage.color import skimage.filters import skimage.util import skimage.segmentation img = skimage.util.img_as_float( plt.imread("d:\lena_std.jpg") ) plt.imshow(img) a = skimage.segmentation.slic(img) plt.imshow( a ) plt.imshow( skimage.segmentation.mark_boundaries(img, a) )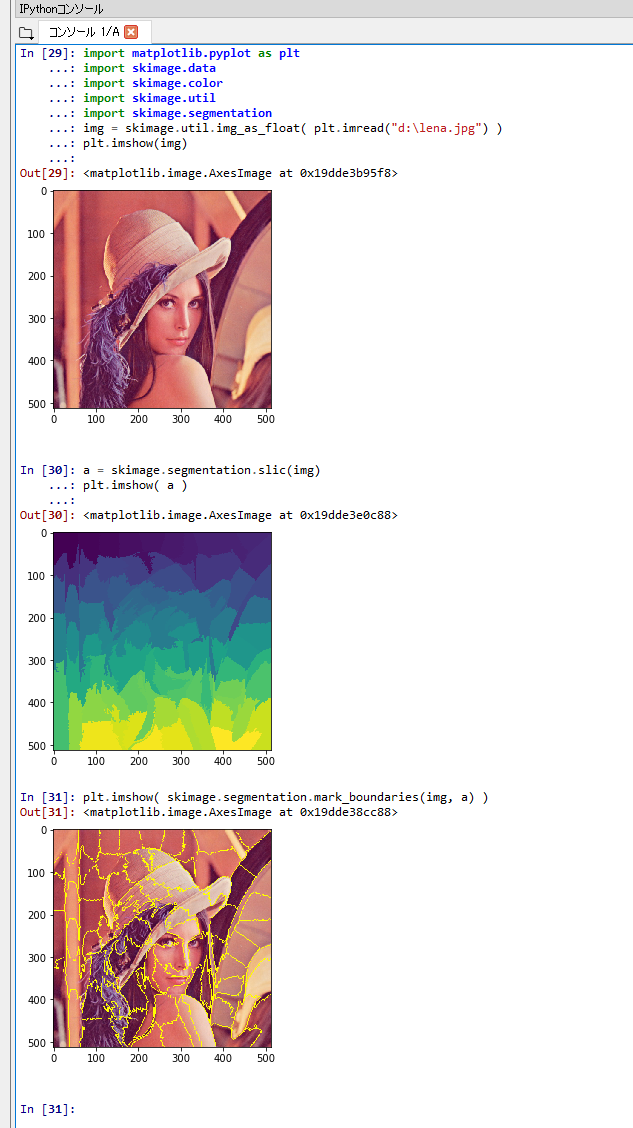
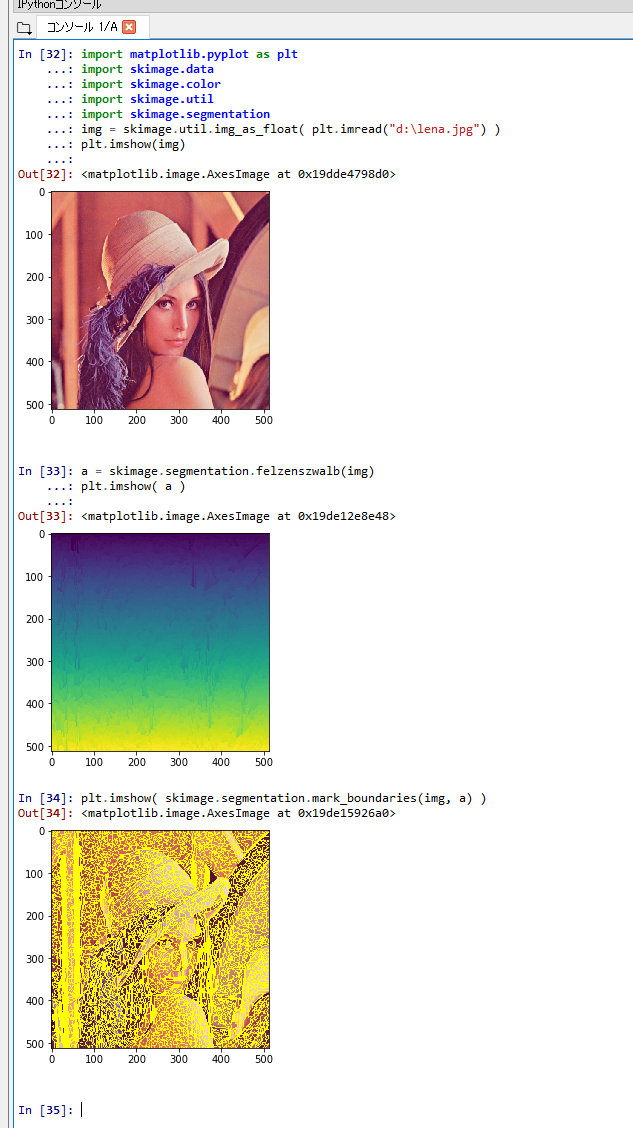
felzenszwalb
felzenszwalb を行う.
Python プログラムを実行する.Matplotlib を使うので,Jupyter QtConsole や Jupyter ノートブック (Jupyter Notebook) の利用が便利である.
%matplotlib inline
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings('ignore') # Suppress Matplotlib warnings
import skimage.data
import skimage.color
import skimage.filters
import skimage.util
import skimage.segmentation
img = skimage.util.img_as_float( plt.imread("d:\lena_std.jpg") )
plt.imshow(img)
a = skimage.segmentation.felzenszwalb(img)
plt.imshow( a )
plt.imshow( skimage.segmentation.mark_boundaries(img, a) )
quickshift
quickshift を行う.
Python プログラムを実行する.Matplotlib を使うので,Jupyter QtConsole や Jupyter ノートブック (Jupyter Notebook) の利用が便利である.
%matplotlib inline
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings('ignore') # Suppress Matplotlib warnings
import skimage.data
import skimage.color
import skimage.filters
import skimage.util
import skimage.segmentation
img = skimage.util.img_as_float( plt.imread("d:\lena_std.jpg") )
plt.imshow(img)
a = skimage.segmentation.quickshift(img)
plt.imshow( a )
plt.imshow( skimage.segmentation.mark_boundaries(img, a) )

watershed
watershed を行う.
Python プログラムを実行する.Matplotlib を使うので,Jupyter QtConsole や Jupyter ノートブック (Jupyter Notebook) の利用が便利である.
%matplotlib inline
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings('ignore') # Suppress Matplotlib warnings
import skimage.data
import skimage.color
import skimage.filters
import skimage.util
import skimage.segmentation
img = skimage.util.img_as_float( plt.imread("d:\lena_std.jpg") )
plt.imshow(img)
a = skimage.segmentation.watershed( skimage.filters.sobel( skimage.color.rgb2gray( img ) ), markers=250 )
plt.imshow( a )
plt.imshow( skimage.segmentation.mark_boundaries(img, a) )

![[kaneko lab.]](https://www.kkaneko.jp/info/logo_png.png)