医用画像診断支援共通プラットフォーム(PLUTO)のインストール,データの読み込み,レンダリング(Windows 上)
優れたソフトウェアであるPLUTOの作者陣に感謝いたします.
- 本Webページは,ソフトウェアPLUTO を利用して得られた.
- ソフトウェアPLUTOの著作権はPLUTOの創作者に帰属する。
PLUTOとは
PLUTOとは、 多臓器・多疾病を対象とした統合型の汎用性の高いコンピュータ支援画像診断システムである。このソフトウェアは文部科学省科学研究費特定領域研究「多次元医用画像の知的診断支援」におけるプロジェクトのひとつとして開発されたものである。 このWebページでは,PLUTOを画像のボリュームレンダリング表示のために用いる.このWebページでは,次の2つについて説明する.
- RGBカラーBMP画像のPLUTO対応形式への変換手順
- 「医用画像診断支援共通プラットフォーム(PLUTO)」を用いた3次元表示
このPLUTOはホームページから入手したアーカイブに改変を加えず、利用許諾契約書を添付することで、ソフトウェアを再配布できる(2010/06時点.必ず,各自で確認すること)。本ソフトウェアを利用して得られた成果物を公表する場合、当該成果物がPLUTOを利用して得られたものであることと、PLUTOの著作権は創作者に帰属することを表示するものとする(これも必ず,各自で確認すること)
- 当該成果物が本ソフトウェアを利用して得られたものであること。
- 本ソフトウェアの著作権は創作者に帰属すること。
PLUTO利用許諾書URL:http://pluto.newves.org/registration/EULA.html
PLUTOのインストール手順
ここではバージョン0.9.8をダウンロードし、インストールを行う手順を示す。
- PLUTO の Web ページを開く
http://pluto.newves.org (現存しない)
- その後,ダウンロードページに進む
- ユーザ登録を行う
メールによりダウンロードページのURLが送られてくる。そのURLよりファイルをダウンロードする。
- URLよりダウンロードしたファイルを,分かりやすいディレクトリに展開することでインストールが完了する。
PLUTOの起動方法
- デスクトップに展開されたディレクトリ内にあるディレクトリ 「x86」 または「x64」を開く
- ファイル「Pluto.exe」を実行する
次のウインドウが立ち上がればPLUTOが正しく実行されている。
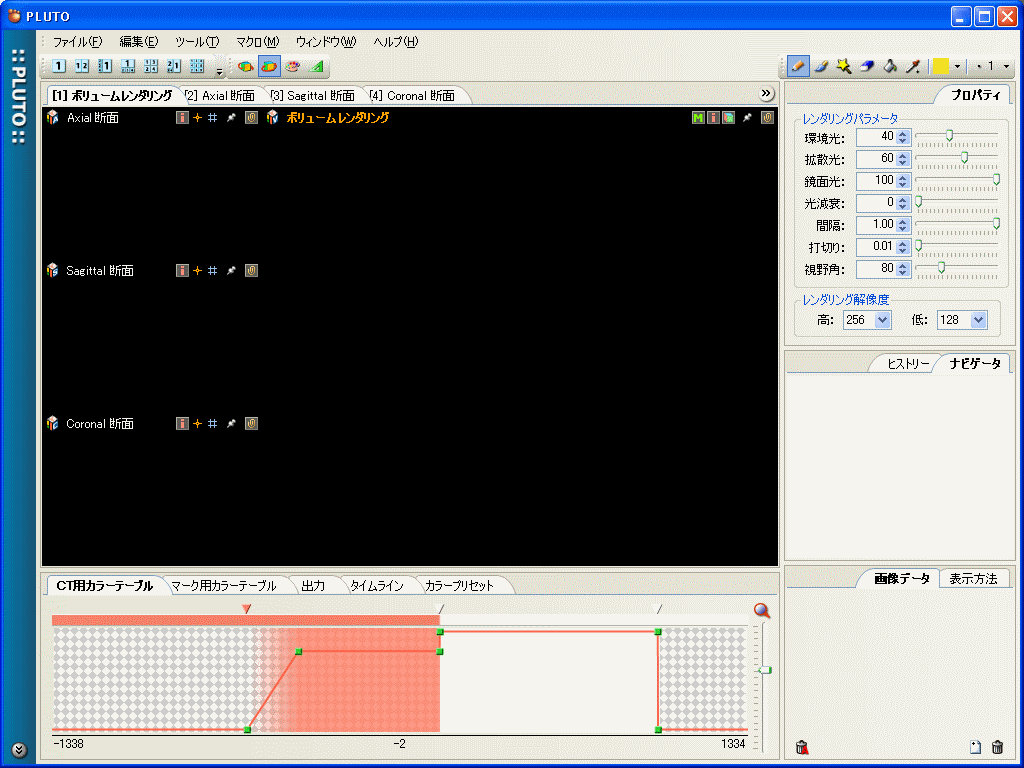
図:PLUTOの実行画面
ここで用いる画像
- CT画像(RGBカラーBMP画像)
- CT画像から臓器領域を抽出したデータ(RGBカラーBMP画像)
- MRA画像(RGBカラーBMP画像)
- MRA画像から血管領域を抽出したデータ(RGBカラーBMP画像)
臓器領域と血管領域は、CT画像とMRA画像の臓器の輪郭を粘性流体位置合わせで補正済みである.
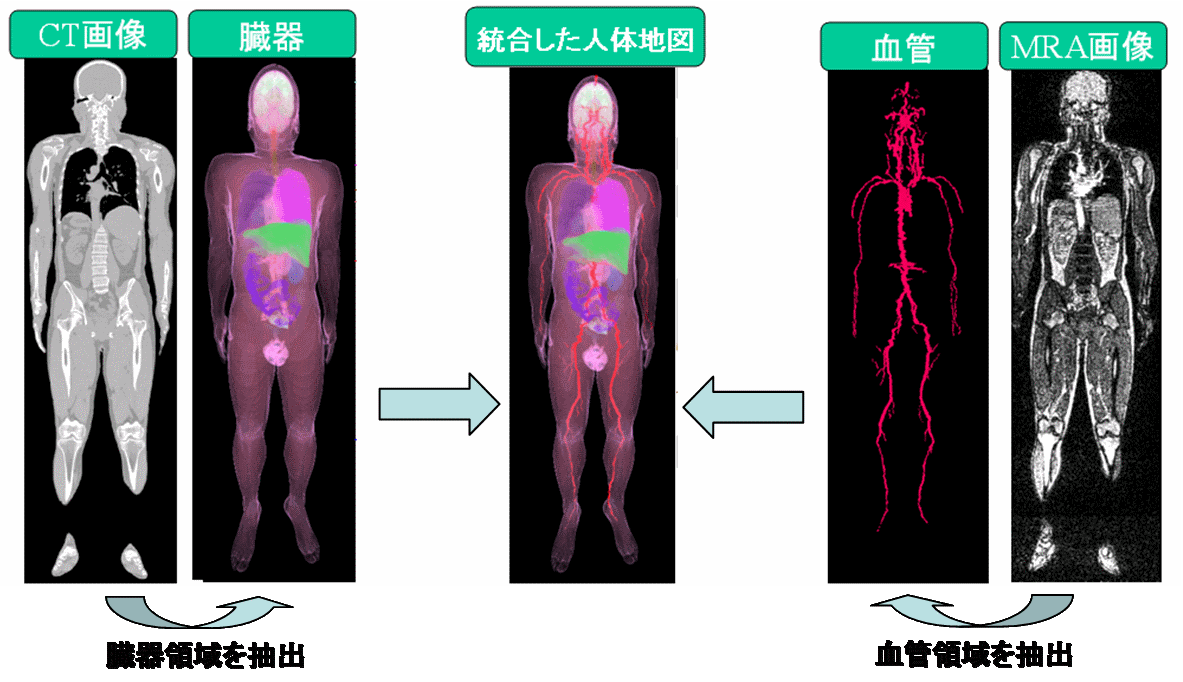
図:臓器と血管の3次元人体地図を統合した3次元人体地図
画像データファイル形式
画像は,4種類ともRGBカラーのBMP形式の断面画像1687枚である.臓器領域,血管領域を抽出したデータも1687枚の断面画像である.これら,臓器領域,血管領域を抽出したデータを重ねあわせて,3次元表示を行うことをPLUTOを用いて行う.
- 臓器領域を抽出したデータ(RGBカラーBMP画像)のディレクトリ:仮にC:/segmentation_data とする
- 血管領域を抽出したデータ(RGBカラーBMP画像)のディレクトリ:仮にC:/vessel とする
RGBカラーBMP画像のPLUTO対応形式への変換手順
PLUTO対応形式には,DICOMとRAW形式がある。ここでは,取り扱いが簡便なRAW形式に変換を行うこととする。RAW形式はヘッダが存在せず、配列に固定長のデータを入れていくものである。
- 臓器領域を抽出したデータ(RGBカラーBMP画像)の,256色グレイスケールBMP画像への変換
まず、臓器領域を抽出したデータ(RGBカラーBMP画像)を,256色グレイスケールBMP画像に変換するプログラムを作成し、カラー画像を256色グレイスケール画像に変換する。なぜ専用のプログラムを作成したかというと、臓器のRGB値を足して3で割る通常のグレイスケール値に変換するものを利用すると、複数の臓器が同じ値に変換されてしまうためである。このプログラムは予め臓器の色を取得しており、その数値を元に、256色中の別々の色に割り当てる。臓器の色は18色あり、それぞれ10、20・・・180までの18通りの色に置き換えている。背景色(R,G,B)=(255,255,255)は190に置き換えている。
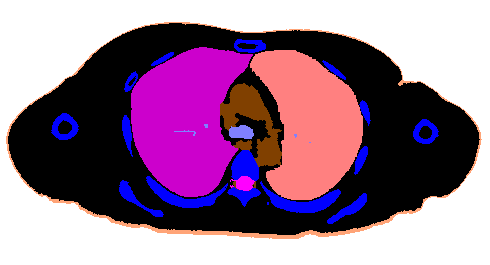

図:変換前のRGBカラーBMP画像と変換後の256色グレイスケールBMP画像clear all; cd "C:/ " %臓器のRGB値 col=[0 0 0;0 0 255;0 128 255;0 151 0;17 177 255;74 255 255;128 0 64;128 64 0;128 128 0;128 128 255;157 255 255;204 0 204;242 0 121;255 0 255;255 47 47;255 74 255;255 128 128;255 164 119;255 255 255]; for i=10:10:1687 file_name=strcat("segmentation_data/zouki_0",dec2base(i,10,4),".bmp"); tmp_=imread(file_name); if(size(size(tmp_))(2)!=3) tmp=zeros(265,490,3); tmp=zeros(265,490,3); else tmp=tmp_; endif tmp2=zeros(265,490); for j=1:490 for k=1:265 for l=1:size(col)(1) if(reshape(tmp(k,j,:),1,3)==col(l,:))tmp2(k,j)=l*10; endif end end end imwrite(tmp2/255,strcat("C:/PLUTO/zouki_gray_0",dec2base(i,10,4),".bmp")); end
カラー画像をグレイスケール画像に変換するプログラム - 臓器領域,血管領域を抽出したデータの統合
次に、臓器領域,血管領域を抽出したデータを統合するプログラムを使う.これは、血管の数値を臓器の画像に上書きするプログラムである。次に、統合したRBGカラーの490x265の画像1687枚を1つの配列の大きさが8bit Unsigned Integerとなるような490x265x1687のRAW形式のデータに変換する。RBGカラーの臓器と血管を統合した画像を256色の色にマッピングを行うOctaveのプログラムを使う.血管の色は200に置き換えている。
この結果,この連番画像は256色グレイスケールBMP画像の連番ファイルであり、臓器の色が10、20、・・・180までと多色で構成されており、血管が200、背景色が190となっている。

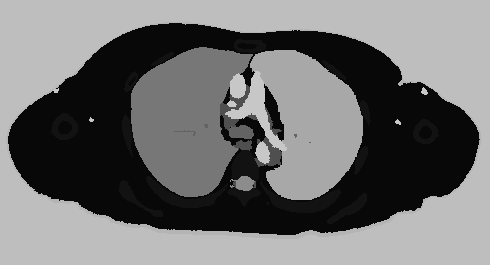
図:結合前の血管画像と、統合した3次元人体地図の画像clear all; cd "C:/ " for i=1:1687 file_name=strcat("PLUTO/zouki_gray_0",dec2base(i,10,4),".bmp"); file_name2=strcat("vessel/out",dec2base(i,10,4),".bmp"); tmp_1=imread(file_name); tmp_2=imread(file_name2); if(size(size(tmp_1))(2)==3) tmp1=tmp_1(:,:,1); else tmp1=tmp_1; endif if(size(size(tmp_2))(2)==3) tmp2=tmp_2(:,:,1); else tmp2=tmp_2; endif tmp2=double(tmp2)*1000+1; tmp3=uint8(tmp1./tmp2); if(max(max(tmp2))>0) tmp1=uint8(tmp2./max(max(tmp2))*200+tmp3; endif imwrite(tmp1,strcat("統合した人体地図/fluid2_",dec2base(i,10,4),".bmp")); end
図:臓器と血管のグレイスケール画像を統合するプログラムそして、臓器と血管を統合した256色グレイスケールBMPを画像変換ソフトBatch goo!を用いて256色tiff形式に変換する。Batch goo!は下記のURLからダウンロードできる。
http://www.vector.co.jp/download/file/win95/art/fh512252.html
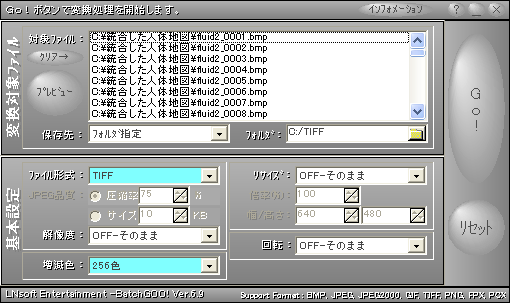
図:Batch goo!の動作画面最後に、MRICROを用いてTIFF画像をアナライズフォーマット(.hdr+.img)形式に変換する。この.imgファイルがPLUTOで読み込むことができるLAW形式のファイルに当たる。MRICROを起動し、Convert Foreign to Analyzeを用いて画像の枚数1687を入力し、下図の手順でTIFFファイルfluid2_0001.tiffを選択し、fluid2_0001.hdrファイルに変換する。
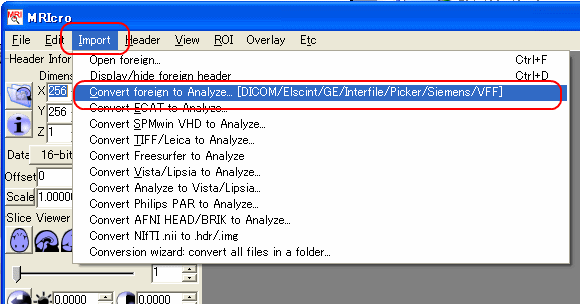
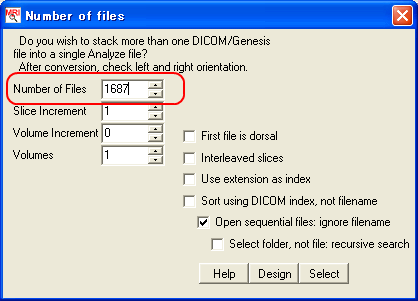
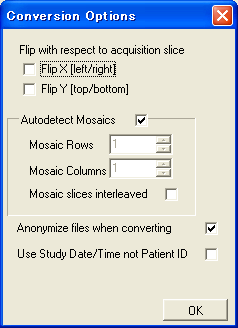
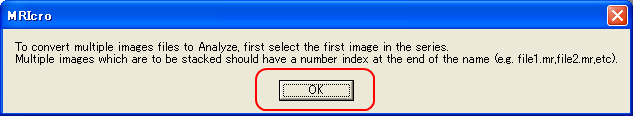
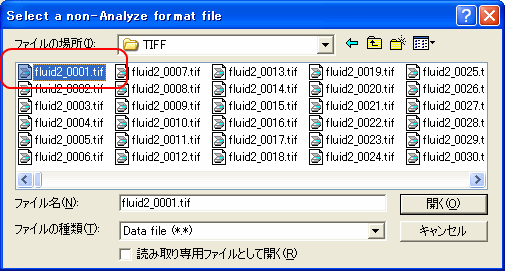
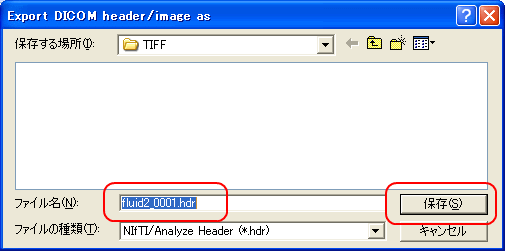
図:MRICROでTIFFファイルを変換する手順
図:MRICROで変換したアナライズフォーマットの画像を縦断面で表示した画像3次元人体地図をPLUTOで読み込ませる
PLUTOで「開く→RAWデータを開く」を選択する。ファイルの種類を「全てのファイル(*.*)」を選び、fluid2_0001.imgファイルを選択する。画像サイズをWidthに490、Heightに265、Depthに1687を設定し、ボクセルサイズをResoXに1を,ResoYに1を,ResoZに0.98を設定し、データタイプにuchar、エンディアンにリトルを設定してOKを押す。
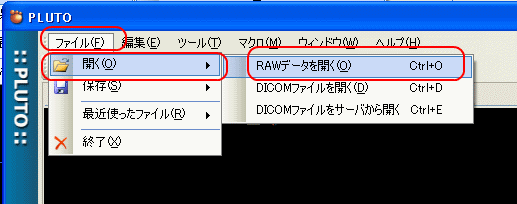
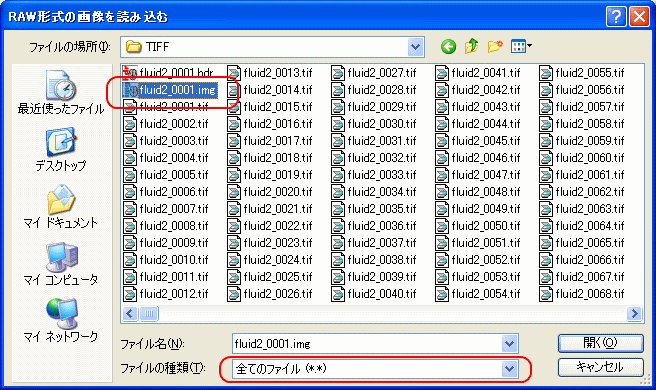
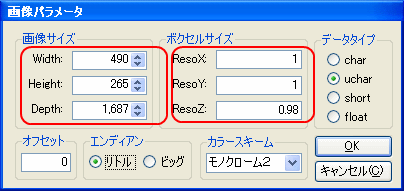
図:PLUTOでRAWデータを開く手順
3次元人体地図の表示の調整(透過度の設定について)
読み込ませただけではカメラ位置が近いため何も分からない画像である。
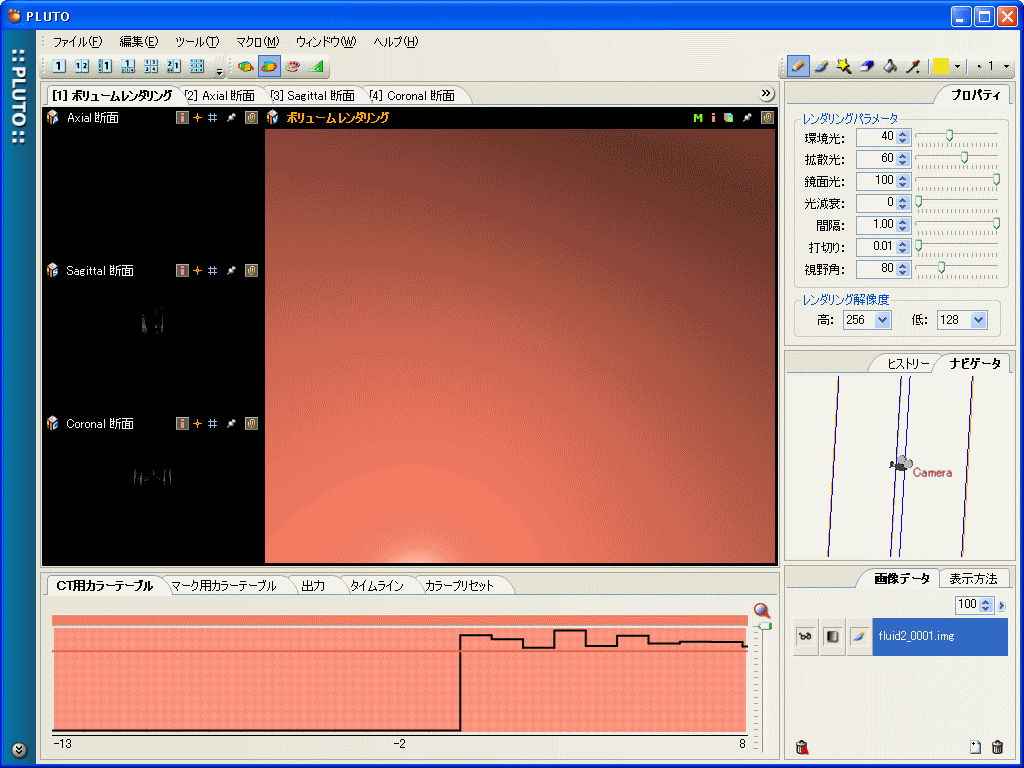
図:読み込ませた後の画像
マウスの右クリックを押しながら右にドラッグすることでカメラを遠ざけることができ、立方体が表示される。左クリックを押しながらドラッグすることで、立方体を回転させることで調節した画像が下図である。
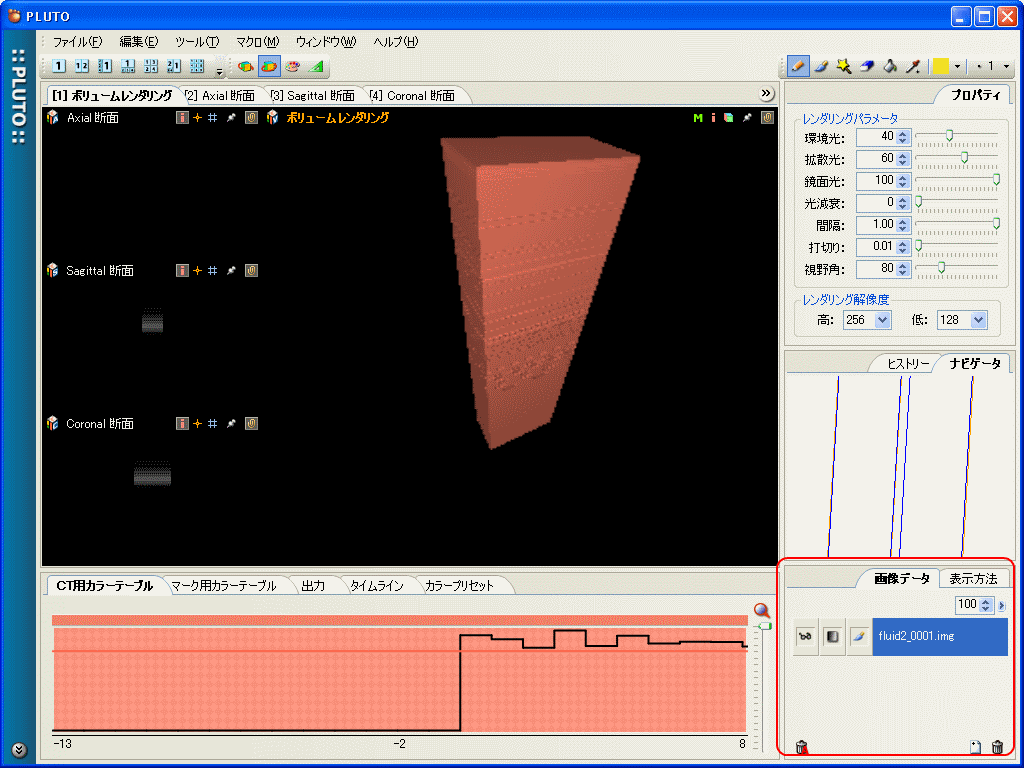
図:カメラを遠ざけ、立方体を回転させ立方体の位置を調節
次に、この画像の描画方法を変更する。CT用カラーテーブルからマーク用カラーテーブルを使用するように設定する。右下の画像データの真ん中のボタンを押し、一番右にある丸いアイコンをクリック.
図:マーク用カラーテーブルを使用するように設定を変更
次に、透過度を設定する。マーク用カラーテーブルをクリック.
図:マーク用カラーテーブルをクリック
そしてマーク用カラーテーブル下のバーを右に伸ばし、0から255まで表示するようにする。
図:255色全てを表示するよう下のバーを右まで伸ばした画像
その後、0にカーソルを合わせて左クリックし、255までドラッグすると、不透明度を変更できるウインドウが表示されるので、不透明度のバーを3%に設定する。

図:全ての色について透明度を設定
そして、背景の値190を無効にする。190の透明度を0にしても同じ画像が得られるため、どちらを行っても良い。
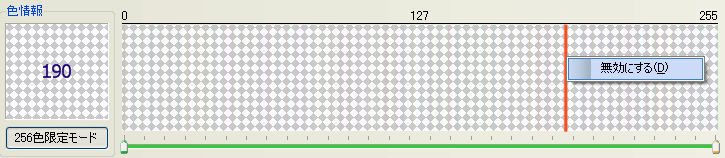
図:背景色を描画しないよう無効にする
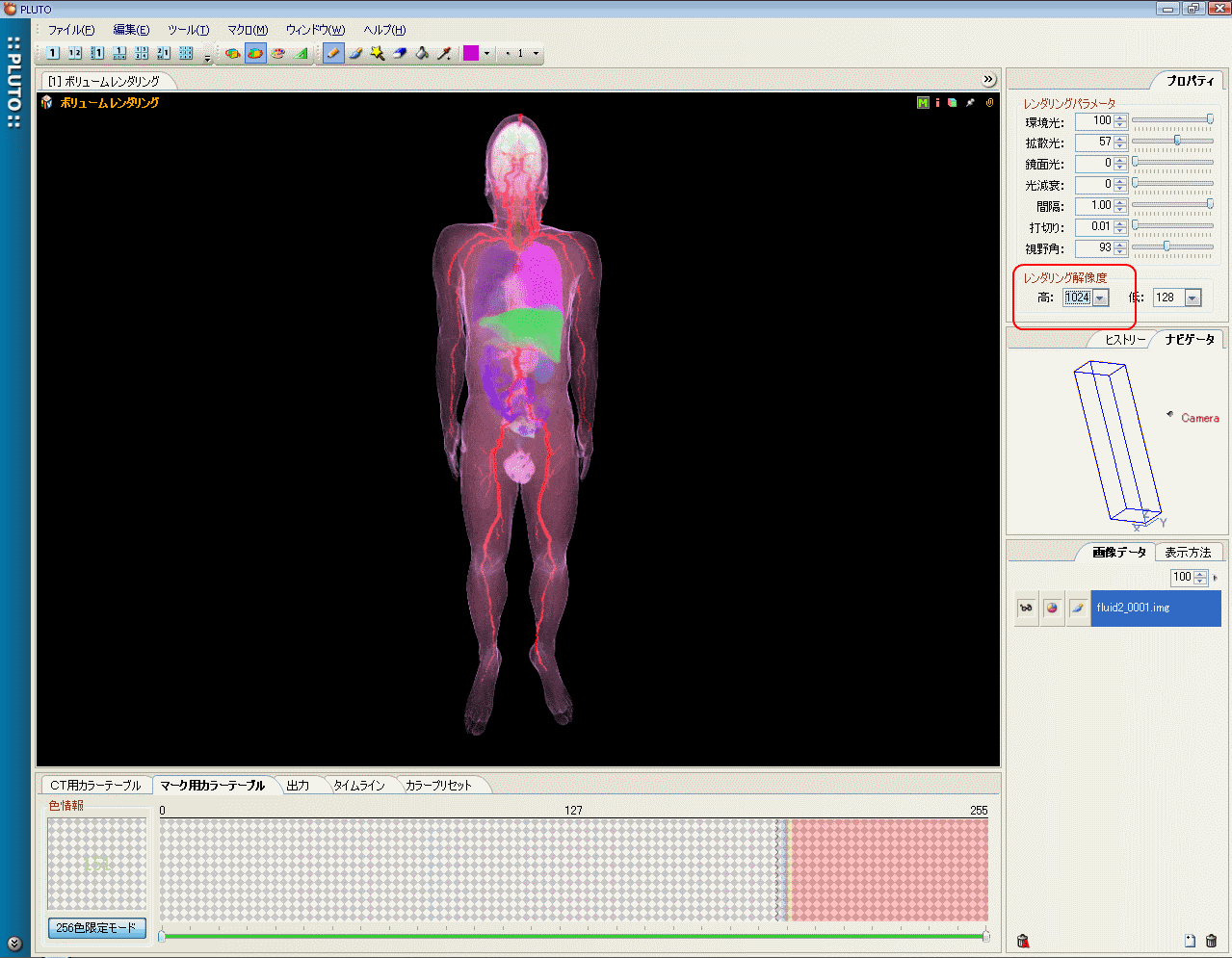
図:3次元人体地図をPLUTOで表示した結果
![[kaneko lab.]](https://www.kkaneko.jp/info/logo_png.png)