主成分分析、ロバスト主成分分析、ICAの実行結果例
事前準備
# 主成分分析
install.packages("pca3d")
# ロバスト主成分分析
install.packages("ppPCA")
# ICA
install.packages("fastICA")
fastICA パッケージの ICA 実行
◆ テストデータ
require(data.table)
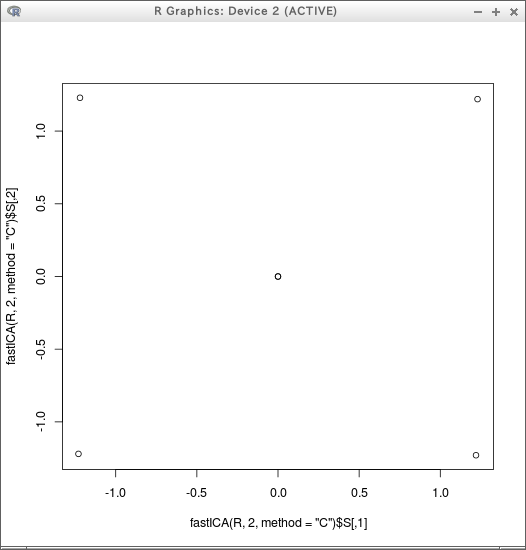
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(fastICA)
plot( fastICA( R, 2, method="C")$S )
◆ Iris データセット
require(fastICA)
plot( fastICA( iris[,c(1:4)], 2, method="C")$S, col=iris[,5] )
◆ ランダム分布データ
require(mvtnorm)
T <- rbind(rmvnorm(200, rep(0, 6), diag(c(5, rep(1,5)))),
rmvnorm( 15, c(0, rep(20, 5)), diag(rep(1, 6))))
require(fastICA)
plot( fastICA( T, 2, method="C")$S, col=(T[,2]>10)+1 )
主成分分析の実行
◆ テストデータ
require(data.table)
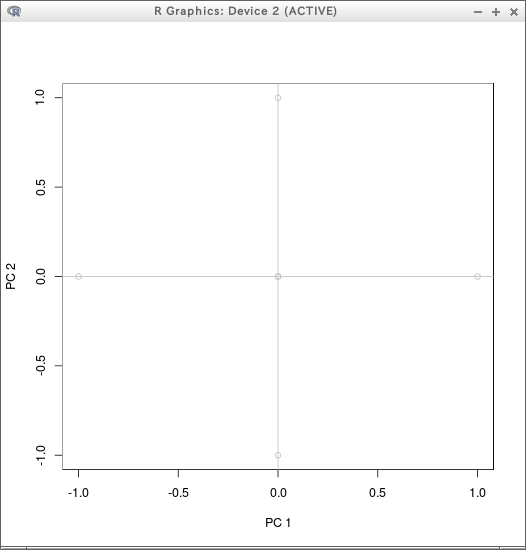
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(pca3d)
pca2d( princomp(R, cor=FALSE) )
require(data.table)
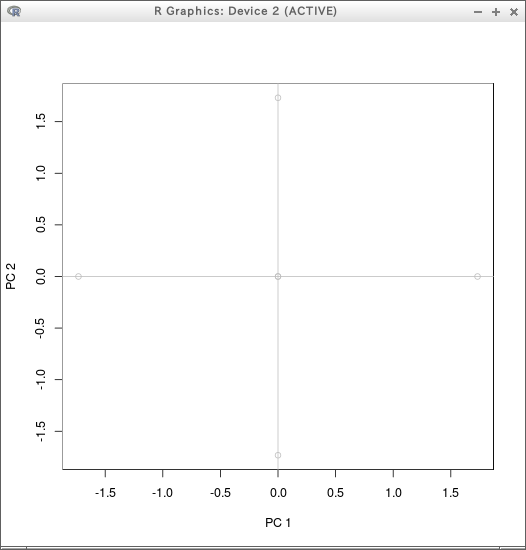
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(pca3d)
pca2d( princomp(R, cor=TRUE) )
require(data.table)
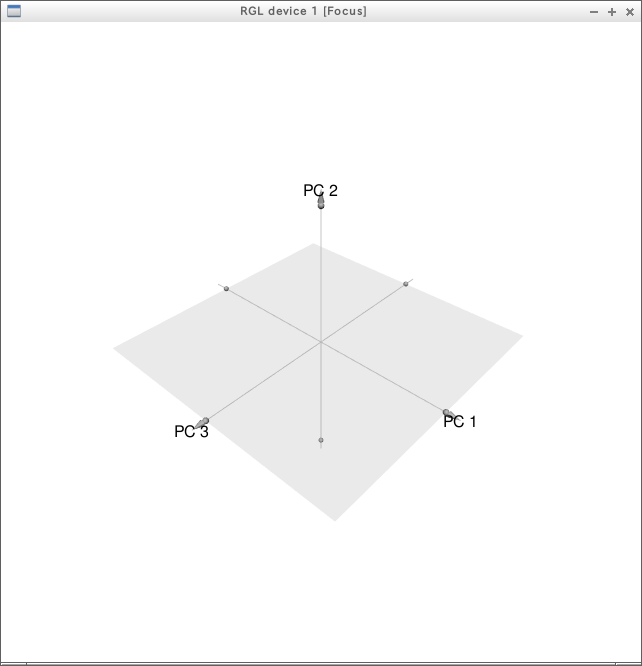
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(pca3d)
pca3d( princomp(R, cor=FALSE) )
require(data.table)
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(pca3d)
pca3d( princomp(R, cor=TRUE) )
◆ Iris データセット
require(pca3d)
pca2d( princomp(iris[,c(1:4)], cor=TRUE), col=iris[,5] )
require(pca3d)
pca3d( princomp(iris[,c(1:4)], cor=TRUE), col=as.numeric(iris[,5]) )
◆ ランダム分布データ
require(mvtnorm)
x <- rbind(rmvnorm(200, rep(0, 6), diag(c(5, rep(1,5)))),
rmvnorm( 15, c(0, rep(20, 5)), diag(rep(1, 6))))
require(pca3d)
pca2d( princomp(x, cor=TRUE), col=(x[,2]>10)+1 )
require(mvtnorm)
x <- rbind(rmvnorm(200, rep(0, 6), diag(c(5, rep(1,5)))),
rmvnorm( 15, c(0, rep(20, 5)), diag(rep(1, 6))))
require(pca3d)
pca3d( princomp(x, cor=TRUE), col=(x[,2]>10)+1 )
ロバスト主成分分析の実行
◆ テストデータ
require(data.table)
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(pca3d)
require(pcaPP)
pca2d( PCAgrid(R, cor=TRUE) )
require(data.table)
R <- data.table( x=c(2,0,1,1,1,1), y=c(1,1,2,0,1,1), z=c(1,1,1,1,2,0) )
require(pca3d)
require(pcaPP)
pca3d( PCAgrid(R, cor=TRUE, k=3 ) )

◆ Iris データセット
require(pca3d)
require(pcaPP)
pca2d( PCAgrid(iris[,c(1:4)], cor=TRUE), col=iris[,5] )
require(pca3d)
require(pcaPP)
pca3d( PCAgrid(iris[,c(1:4)], cor=TRUE, k=3), col=as.numeric(iris[,5]) )
◆ ランダム分布データ
require(mvtnorm)
x <- rbind(rmvnorm(200, rep(0, 6), diag(c(5, rep(1,5)))),
rmvnorm( 15, c(0, rep(20, 5)), diag(rep(1, 6))))
require(pca3d)
require(pcaPP)
pca2d( PCAgrid(x, cor=TRUE), col=(x[,2]>10)+1 )
require(mvtnorm)
x <- rbind(rmvnorm(200, rep(0, 6), diag(c(5, rep(1,5)))),
rmvnorm( 15, c(0, rep(20, 5)), diag(rep(1, 6))))
require(pca3d)
require(pcaPP)
pca3d( PCAgrid(x, cor=TRUE, k=3), col=(x[,2]>10)+1 )
(参考) plot( iris[,c(1:2)], col=iris[,5] ) の実行結果例
![[kaneko lab.]](https://www.kkaneko.jp/info/logo_png.png)